- Projekte
- > Ausgezeichnet!
- > OvG-Preis
- > Preisträger 2005
Preisträger 2005: Fingerabdruck überführt Mikroben: Identifizierung von schädlichen Mikroorganismen"

Bisher war die Identifizierung von verderbniserregenden Mikroorganismen sehr kostspielig und aufwändig. In mehreren Forschungs- vorhaben sollte daher eine neue Technik entwickelt werden, die folgende Kriterien erfüllt: einfache Anwendung, schnelles und sicheres Ergebnis, hoher Probendurchsatz sowie niedrige Kosten für Verbrauchsmaterial.
Das ist Prof. Siegfried Scherer und Dr. Herbert Seiler vom Zentralinstitut für Ernährungs- und Lebensmittelforschung der Technischen Universität München gelungen, die für diese Arbeiten mit dem Otto-von-Guericke-Preis 2005 der AiF ausgezeichnet wurden. Der Preis würdigt herausragende Leistungen auf dem Gebiet der Industriellen Gemeinschaftsforschung (IGF).
Fingerabdrücke von Bakterien
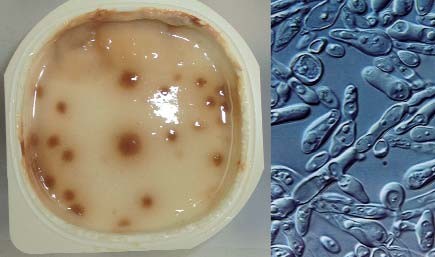
Der Lösungsansatz des Forscherteams besteht in einer Weiterentwicklung der so genannten FT-IR-makrospektroskopischen Charakterisierung von Mikroben. Das Verfahrensprinzip ist einfach: Von der getrockneten Suspension der Reinkultur eines unbekannten Mikroorganismus wird ein Infrarot-Spektrum aufgenommen, welches einen individuellen Fingerabdruck der Art darstellt. Dieses Spektrum wird mit Spektren bekannter Mikroben verglichen, die in Referenzspektrenbibliotheken abgelegt sind. Die Forscher haben das Verfahren in mehreren IGF-Projekten für lebensmittelbürtige Keime etabliert und für den Einsatz in der Lebensmittelindustrie adaptiert.
Erkennungsdienstliche Erfassung

Identifizierung im Turbobetrieb

Lernen vom Gehirn
Um das Verfahren direkt in Lebensmittel verarbeitenden Betrieben einfach und sicher einsetzen zu können, äußerten Industrievertreter in den Projektbegleitenden Ausschüssen immer wieder den Wunsch nach einer weitgehend automatisierten Auswertung der Spektren. Eine solche Möglichkeit wird im Rahmen eines weiteren IGF-Vorhabens untersucht. Ermöglicht werden soll diese Weiterentwicklung durch den Einsatz künstlicher neuronaler Netze, die in anderen Bereichen bereits zur Datenanalyse genutzt werden. Deren Aufbau ist dem Nervensystem von Lebewesen nachempfunden. Durch die Anwendung dieser Technik können die Auswertung vereinfacht und die Identifikationsgüte verbessert werden, weil sehr viel mehr Information als bisher aus den Spektren extrahiert werden kann. Die bisherigen Ergebnisse lassen eine weitere Steigerung der Leistungsfähigkeit und Anwendbarkeit der FT-IR-Technologie erwarten. Außerdem zeichnet sich in der Identifizierungsgüte von sehr ähnlichen Arten sogar eine unerwartet deutliche Verbesserung ab.
Technologietransfer
Der Transfer der Ergebnisse in die Industrie erfolgte in zahlreichen Sitzungen der Projektbegleitenden Ausschüsse, in denen Industrievertreter aus verschiedenen Unternehmen kontinuierlich mitarbeiteten. Das Verfahren ist inzwischen in mehreren Laboren lebensmittelverarbeitender Betriebe sowie in zwei mittelständischen und einem staatlichen Untersuchungslabor etabliert, die die Analyse von Verderbniserregern als Dienstleistung anbieten. Gespräche mit weiteren Interessenten aus der Industrie sind geplant und auch das internationale Marktpotenzial ist groß, was nicht zuletzt durch Studienaufenthalte von Gastwissenschaftlern ausländischer Universitäten und Unternehmen an der TU München deutlich wurde. Eine weitere Anwendungsmöglichkeit der neuen FT-IR-Technologie zeichnet sich außerdem in der Qualitätssicherung von Pharmabetrieben ab.
Projektbeteiligte
Forschungsstelle:
(Stand: September 2005)
... ein Projekt der Industriellen Gemeinschaftsforschung (IGF)